Estudio comparativo de modelos de aprendizaje profundo para segmentar tejido adiposo abdominal en tomografía axial computarizada
DOI:
https://doi.org/10.53903/01212095.280Palabras clave:
Grasa abdominal, Grasa intraabdominal, Tomografía computarizada por rayos XResumen
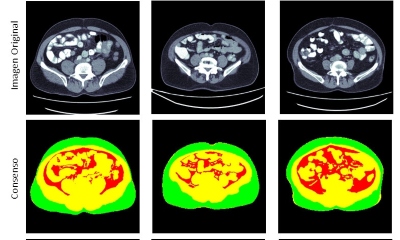
Propósito: El análisis de composición corporal sirve como indicador de ciertas condiciones médicas como el síndrome metabólico, el cáncer, la diabetes o las enfermedades cardiovasculares. Tradicionalmente, estos análisis se realizan mediante métodos antropométricos o herramientas clínicas que proporcionan un resultado aproximado. Usando la familia de arquitecturas de Aprendizaje Profundo U-NET, se realizó una segmentación completamente automática del tejido adiposo abdominal visceral y subcutáneo. Se estudiaron estos resultados de segmentación y se compararon con el patrón de oro generado por segmentación manual de expertos. Materiales y métodos: Se emplearon cuatro variaciones de la arquitectura de Aprendizaje Profundo de U-Net: U-Net, R2U-Net, Attention U-Net y Attention R2U-Net. Estos métodos se entrenaron en un conjunto de datos que consta de 554 imágenes recopiladas entre 2015 y 2017 en el Hospital Universitario San Ignacio y en el Instituto IDIME en Bogotá, Colombia. Esta base de imágenes contiene anotaciones para tres tejidos diferentes: grasa visceral, grasa subcutánea y otros tejidos, generadas a través de herramientas de segmentación semiautomáticas. Resultados: El índice de Sørensen-Dice se utiliza como métrica de evaluación al comparar con los datos obtenidos del patrón de oro, que consiste en segmentaciones manuales realizadas por expertos. Se obtuvo que la arquitectura U-Net fue la más precisa en términos de segmentación de la composición corporal general, con un puntaje promedio de Dice de 93,0 %, seguida de cerca por la arquitectura Attention U-Net con un puntaje promedio de Dice de 92,0 %. Conclusiones: Según los resultados, se descubrió que las arquitecturas U-Net y Attention U-Net son las más adecuadas para el análisis de la composición corporal. Los resultados de segmentación producidos por estos métodos podrían usarse para obtener métricas precisas y ayudar a los médicos a comprender la condición física del paciente.
Descargas
Referencias bibliográficas
Afshin A, Vos T, Murray C, Fernandes J, Silverberg J, Bjertness E, et al. Health effects of overweight and obesity in 195 countries over 25 years. New Eng J Med. 2017;377: 13-27
González MC, Pastore C, Orlandi S, Heymsfield S. Obesity paradox in cancer: New insights provided by body composition. Am J Clin Nut. 2014;99
Seabolt LA, Welch EB, Silver HJ. Imaging methods for analyzing body composi¬tion in human obesity and cardiometabolic disease. Ann New York Acad Sci. 2015;1353:41-59
Ronneberger O, Fischer P, Brox T. U-Net: convolutional networks for biomedical image segmentation [Presentación]. En: Computación de imágenes médicas e intervención asistida por computadora (MICCAI). 2015. Disponible en: https://link.springer.com/chapter/10.1007/978-3-319-24574-4_28
Lee S, Liu J, Yao J, Kanarek A, Summers R, Pickhardt P. Fully automated segmentation and quantification of visceral and subcutaneous fat at abdominal CT: Application to a longitudinal adult screening cohort. Br J Radiol. 2018;91:20170968
Hui SCN, Zhang T, Shi L, Wang D, Ip CB, Chu WCW. Automated segmentation of abdominal subcutaneous adipose tissue and visceral adipose tissue in obese adolescent in MRI. Magn Resonan Imag. 2018;45:97-104
Nemoto M, Yeernuer T, Masutani Y, Nomura Y, Hanaoka S, Miki S, et al. Develop¬ment of automatic visceral fat volume calculation software for CT volume data. J Obesity. 2014;2014:495084
Wang Z, Hounye A, Zhang J, Hou M, Qi M. Deep learning for abdominal adipose tissue segmentation with few labelled samples. International J Comp Assis Radiol Surg. 2021;17
Micomyiza C, Zou B, Li Y. An effective automatic segmentation of abdominal adipose tissue using a convolution neural network. Diabetes & Metabolic Syndrome: Clinical Research & Reviews. 2022;16:102589
Li H, Luo H, Liu Y. Paraspinal muscle segmentation based on deep neural network. Sensors. 2019;19:2650
Estrada S, Lu R, Conjeti S, Orozco-Ruiz X, Panos-Willuhn J, Breteler MMB, et al. FatSegNet: A fully automated deep learning pipeline for adipose tissue segmentation on abdominal dixon MRI. Magn Reson Med. 2020;83:1471-83
Alom MZ, Hasan M, Yakopcic C, Taha TM, Asari VK. Recurrent residual convolu¬tional neural network based on U-Net (R2U-Net) for medical image segmentation [internet]. 2018 [citado: 2023 feb. 15]. Disponible en: https://arxiv.org/abs/1802.06955
Oktay O, Schlemper J, Folgoc LL, Lee M, Heinrich M, Misawa K, et al. Attention U-Net: learning where to look for the pancreas [internet]. 2018 [citado: 2023 feb. 15]. Disponible en: https://arxiv.org/abs/1804.03999
Zhang L, Zuo Q, Chen S, Wang Z. R2AU-Net: Attention recurrent residual convo¬lutional neural network for multimodal medical image segmentation. Sec Commun Net. 2021;2021:6625688
Weston AD, Korfiatis P, Kline TL, Philbrick KA, Kostandy P, Sakinis T, et al. Auto¬mated abdominal segmentation of CT scans for body composition analysis using deep learning. Radiology. 2019;290:669-79
Dabiri S, Popuri K, Cespedes E, Caan B, Baracos V, Beg MF. Deep learning method for localization and segmentation of abdominal CT. Comput Med Imag Graph. 2020;85:101776
Hemke R, Buckless C, Tsao A, Wang B, Torriani M. Deep learning for automated segmentation of pelvic muscles, fat, and bone from CT studies for body composition assessment. Skel Radiol. 2019;49
Koitka S, Kroll L, Malamutmann E, Oezcelik A, Nensa F. Fully automated body composition analysis in routine CT imaging using 3D semantic segmentation convo¬lutional neural networks. Eur Radiol. 2020;31.
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2024 Revista colombiana de radiología

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-CompartirIgual 4.0.
La Revista Colombiana de Radiología es de acceso abierto y todos sus artículos se encuentran libre y completamente disponibles en línea para todo público sin costo alguno.
Los derechos patrimoniales de autor de los textos y de las imágenes del artículo como han sido transferidos pertenecen a la Asociación Colombiana de Radiología (ACR). Por tanto para su reproducción es necesario solicitar permisos y se debe hacer referencia al artículo de la Revista Colombiana de Radiología en las presentaciones o artículos nuevos donde se incluyan.